酸化ストレス応答に着目したデータベース「Localizatome」を開発
8,000種超のタンパク質局在変化を網羅的解析、老化・がんなどの研究や創薬に貢献
ポイント
- 酸化ストレス条件下におけるタンパク質局在の変化を網羅的に解析した、世界初のデータベース「Localizatome(ローカライゼトーム)」を開発
- ヒト遺伝子ライブラリー、独自のハイスループット顕微鏡システム、機械学習を組み合わせ、生細胞内のストレス応答をリアルタイムで解析
- 老化やがんなど酸化ストレス関連疾患の分子メカニズムの解明や、新たな治療法開発への貢献が期待される
概要
東京科学大学(Science Tokyo)大学院医歯学総合研究科 システム発生再生医学分野の松島隆英助教および淺原弘嗣教授を中心に、大阪大学、理化学研究所、武蔵野大学、産業総合研究所の研究チームは、酸化ストレス[用語1]に応答して細胞内局在が変化するタンパク質を網羅的に解析した、世界初のデータベース「Localizatome(ローカライゼトーム)」を開発しました。
本研究では、ハイスループット顕微鏡システム[用語2]と機械学習[用語3]を駆使して、生細胞内の8,055種類のヒトタンパク質の局在情報を収集し、そのうち1,910種類が酸化ストレス依存的に集積体を形成することを発見しました。
酸化ストレスは老化やがんなど、さまざまな疾患の発症および進行に関わる重要な要因であり、「Localizatome」は、ストレス応答時におけるタンパク質の動態を理解するための貴重なリソースとなります。特に本研究では、タンパク質の集積に関わる特定のアミノ酸配列モチーフを同定しており、今後の創薬研究やストレス関連疾患の治療法開発への貢献が期待されます。
さらに、本研究の手法は、酸化ストレス以外のさまざまな生物学的プロセスにおけるタンパク質動態の解析にも応用可能です。
本成果は、4月23日(現地時間)付で、オンラインデータベースジャーナル「Database」に掲載されました。
本研究成果のデータベース URL: https://localizatome.embrys.jp/
背景
タンパク質の細胞内局在は、その機能を理解するうえで重要です。さまざまなストレス条件下では、多くのタンパク質が動的に局在を変化させることが知られています。
既存のデータベースは、主に定常状態における局在情報を提供していますが、ストレス応答時における動的な局在変化に関する情報は限られていました[参考文献1-3]。
なかでも酸化ストレスは、老化やがんをはじめとする多くの疾患に関与しており、タンパク質の構造変化や集積を引き起こすことが知られています。
しかし、酸化ストレス下におけるタンパク質の局在変化を網羅的に解析したデータベースは、これまで存在していませんでした。
研究成果
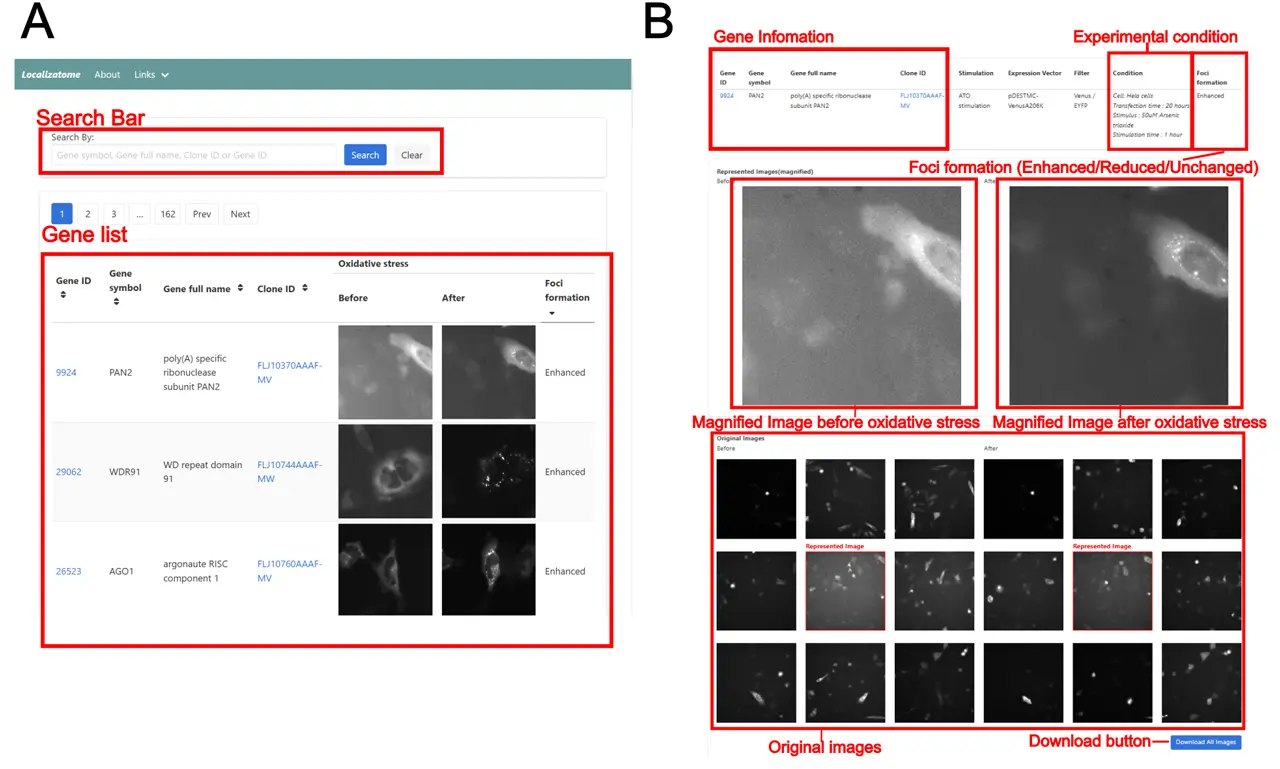
東京科学大学の研究チームは、酸化ストレス応答時におけるタンパク質の局在変化を網羅的に解析した、世界初のデータベース「Localizatome(ローカライゼトーム)」を開発しました(図1)。
蛍光タンパク質融合ヒト遺伝子ライブラリー、独自開発の顕微鏡システム、機械学習アルゴリズムを組み合わせることで、8,055種類のヒトタンパク質について、信頼性の高い局在情報を提供しています。
その結果、1,910種類のタンパク質が酸化ストレス依存的に顕著な局在変化、特に集積体の形成を示すことを明らかにしました。さらに、これらの集積体形成タンパク質を解析することで、特定のアミノ酸の繰り返し配列や特異的なモチーフを同定しました(図2)。
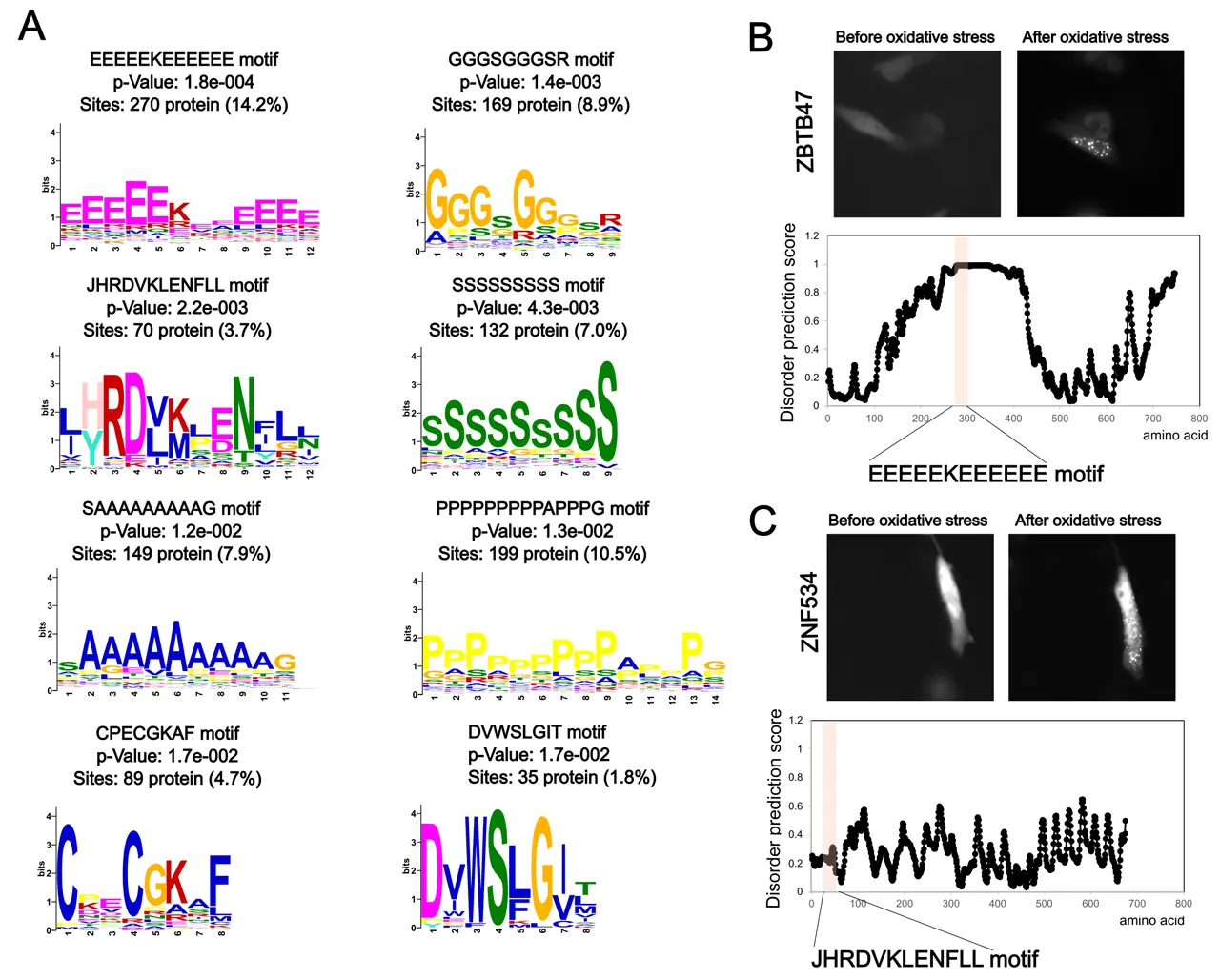
社会的インパクト
「Localizatome」は、酸化ストレス関連疾患の分子メカニズム解明に貢献する、重要なリソースとなります。
本研究では、ストレス顆粒やオートファジー関連タンパク質などの既知のストレス応答性タンパク質に加え、Hippoシグナル経路、細胞分裂、タンパク質分解に関わる多くのタンパク質が、酸化ストレス依存的に局在を変化させることが明らかになりました。
これらの知見は、酸化ストレスによって引き起こされる細胞周期停止などの細胞応答機構の理解を深め、老化やがんなどの疾患メカニズムの解明や、バイオマーカーの探索に貢献すると期待されます。
さらに、タンパク質の集積に関与する新たなモチーフやドメインの発見は、創薬研究や治療法の開発へとつながる可能性を秘めています。
今後の展開
「Localizatome」データベースは現在、主に酸化ストレス依存的なタンパク質の集積体形成に焦点を当てていますが、今後は酸化ストレス以外の重要な生物学的プロセス(細胞分化、飢餓、小胞体ストレスなど)におけるタンパク質動態についても、系統的なデータベースの構築を進めていく予定です。
さらに、既存の遺伝子発現データベースやプロテオミクスデータベースと「Localizatome」の情報を統合することにより、新たな機能性タンパク質ドメインや細胞構造の発見へとつながることが期待されます。
付記
本研究は、科学研究費助成事業(17K15447, 26113008, 20H05696)と日本医療研究開発機構(JP20gm0810008、JP23gm0010009)の支援を受けて行われました。
参考文献
- [1]
- Cho NH, Cheveralls KC, Brunner A-D et al. OpenCell: endogenous tagging for the cartography of human cellular organization. Science 2022;375:eabi6983.
- [2]
- Maruyama Y, Kawamura Y, Nishikawa T et al. HGPD: human Gene and Protein Database, 2012 update. Nucleic Acids Res 2012;40:D924-9.
- [3]
- Thul PJ, Åkesson L, Wiking M et al. A subcellular map of the human proteome. Science 2017;356:eaal3321.
用語説明
- [用語1]
- 酸化ストレス:体内で発生する活性酸素種(スーパーオキシド、過酸化水素など)が過剰に蓄積した状態。通常の細胞機能を妨げ、タンパク質や遺伝子などさまざまな分子に傷害を与え、老化や癌などの疾患の原因となる。
- [用語2]
- ハイスループット顕微鏡システム:多数の試料を自動的に撮影できる顕微鏡システム。本研究では、ロボットによる試料搬送機能を備え、数百枚の培養プレートを自動観察できる特殊な装置を浜松ホトニクス製顕微鏡システムに組み込んだ独自のシステムを使用。
- [用語3]
- 機械学習:コンピュータがデータからパターンを学習し、判断や予測を行う技術。本研究では、画像からタンパク質の集積体を自動的に検出・評価するために使用された。
論文情報
- 掲載誌:
- DATABASE
- タイトル:
- Localizatome: a database for stress-dependent subcellular localization changes in proteins
- 著者:
- Localizatome: A Database for Stress-Dependent Subcellular Localization Changes in Proteins
研究者プロフィール
松島 隆英 Takahide MATSUSHIMA
東京科学大学 大学院医歯学総合研究科 システム発生再生医学 助教
研究分野:分子生物学、発生学
淺原 弘嗣 Hiroshi ASAHARA
東京科学大学 大学院医歯学総合研究科 システム発生再生医学 教授
研究分野:分子生物学(遺伝子発現)、発生・再生医学、整形外科学、リウマチ学
関連リンク
お問い合わせ
東京科学大学 大学院医歯学総合研究科 システム発生再生医学
教授 淺原 弘嗣
- Tel
- 03-5803-5015
- Fax
- 03-5803-5810
- asahara.syst@tmd.ac.jp