要点
- 乳癌幹細胞様集団に特異的に発現し乳癌幹細胞性を制御するRNA結合タンパク質としてZCCHC24を発見しました。
- ZCCHC24は乳癌幹細胞性を特徴づける遺伝子群のmRNAの非翻訳領域に存在する"UGUWHWWA"という配列を認識しmRNAを安定化する新規制御メカニズムを示しました。
- ZCCHC24の標的の一つの転写因子ZEB1はZCCHC24の発現を転写調節しており、転写後調節と転写調節とのポジティブフィードバック機構を介して乳癌幹細胞性を獲得することを明らかにしました。
- ZCCHC24-ZEB1フィードバックを標的にしたトリプルネガティブ乳癌(TNBC)の病態解明と新規治療戦略への応用が期待されます。
概要
東京科学大学(Science Tokyo)※ 大学院医歯学総合研究科 システム発生・再生医学分野の淺原弘嗣教授、内田雄太郎博士研究員・臨床研修医らの研究グループは、金沢大学 がん進展制御研究所の後藤典子教授との共同研究で、難治性乳癌サブタイプの一つであるトリプルネガティブ乳癌(TNBC)[用語1]の癌幹細胞性がRNA結合タンパク質ZCCHC24の転写後調節機構により制御されることを突き止めました。この研究は米国国立衛生研究所、国立研究開発法人日本医療研究開発機構(AMED)の革新的先端研究開発支援事業(AMED-LEAP)、文部科学省科学研究費補助金、ならびに金沢大学がん進展制御研究所共同研究費の支援のもとで行われ、その研究成果は、国際科学誌EMBO Reportsに、2024年10月17日午前10時(ロンドン時間)にオンライン版で発表されました。
- 2024年10月1日に東京医科歯科大学と東京工業大学が統合し、東京科学大学(Science Tokyo)となりました。
背景
乳癌は日本で年間9万人以上が罹患する、女性において最も頻度が高い癌種です。中でもトリプルネガティブ乳癌(TNBC)は乳癌全体の約20%を占め、他のサブタイプと異なり十分な治療法が確立されていないため、予後不良の疾患として知られています。TNBCにおいてはCD44陽性CD24陰性などの細胞膜上のマーカーで特徴づけられ、化学療法に対する治療抵抗性や乳癌の再発、腫瘍形成能に寄与する細胞集団は乳癌幹細胞と呼ばれ、転写制御や転写後制御等による遺伝子発現制御機構により、その幹細胞性が獲得されると考えられてきましたが、その詳細な分子機構はこれまで明らかではありませんでした。
転写後制御をつかさどる代表的なタンパク質としてRNA結合タンパク質が知られています。RNA結合タンパク質は標的遺伝子のmRNA上のcis-element[用語2]と呼ばれる配列に直接結合することで、翻訳の亢進・減衰、およびmRNAの安定化・分解を介して発現制御を行うことで、標的遺伝子の安定的な発現・細胞の恒常性維持に対して非常に重要な役割を果たします。これまで乳癌以外のいくつかの癌種ではRNA結合タンパク質による癌細胞の制御機構は知られていましたが、TNBCにおけるRNA結合タンパク質を介した癌幹細胞性の制御機構は全く不明でした。
研究成果
本研究グループはTNBC患者由来検体に対してシングルセルRNAシークエンス解析[用語3]を行い、乳癌幹細胞を含む間葉系細胞集団に特異的に発現する遺伝子を探索し、RNA結合タンパク質ZCCHC24を同定しました。これまで報告がほとんどされてこなかったZCCHC24分子の特性や遺伝子発現制御機構を解析するため、RNAシークエンスを用いた遺伝子発現解析、BRICシークエンス[用語4]による網羅的なmRNAの安定性解析、およびPAR-CLIP法[用語5]を用いたZCCHC24の結合標的の網羅的解析を行いました。その結果として、ZCCHC24がZEB1, NOTCH2, NRP1, CD44といった乳癌幹細胞性獲得のために重要な遺伝子群のmRNA非翻訳領域に特異的に存在する塩基配列"UGUWHWWA"というcis-elementを認識して結合することで、これらの遺伝子の発現を上昇させて乳癌幹細胞性を獲得することが明らかになりました(図1)。
さらにZCCHC24の発現を制御する上流転写因子の同定をTNBC細胞に対するHi-C[用語6]やChIPシークエンス[用語7]によりデータ解析を行ったところ、ZCCHC24の標的因子であり乳癌幹細胞性を制御することが報告されている転写因子ZEB1がZCCHC24の発現を転写活性化させることが明らかとなり、転写因子ZEB1とRNA結合タンパク質ZCCHC24との間で転写調節と転写後調節によるポジティブフィードバックの形成が確認されました。
次に、ZCCHC24の腫瘍形成における機能解析を進め、ZCCHC24の発現を減少させたTNBC患者由来検体においてはin vivoレベルで腫瘍形成能が減弱することを確認した。さらに、形成された腫瘍に対するシングルセルRNAシーケンス解析を用い、ZCCHC24の発現減少による乳癌幹細胞を含む間葉系細胞集団の大きく減少していることを明らかにしました(図2)。
最後にTNBC患者由来検体を皮下移植したマウスにZCCHC24に対するsiRNA製剤とTNBCに対する使用が有望視される化合物であるBET阻害剤 JQ1とを併用投与すると腫瘍の成長が相加的に抑制されることを明らかにしました(図3)。
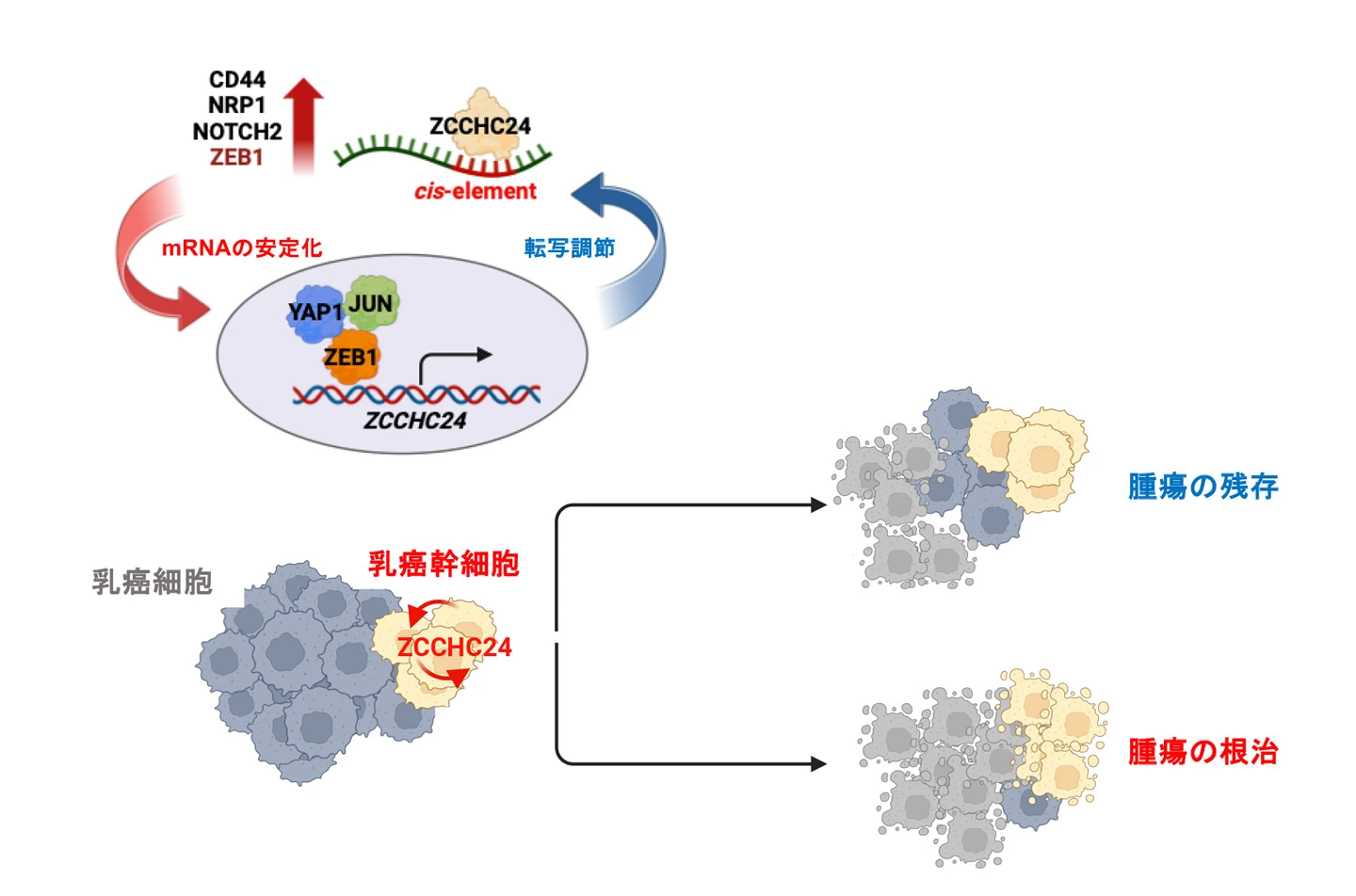
これらの結果から、ZCCHC24によるmRNAの安定化を介した転写後調節制御とZEB1による転写調節制御が、互いにポジティブフィードバックを形成することでTNBCの癌幹細胞性が維持されることを明らかにしました。
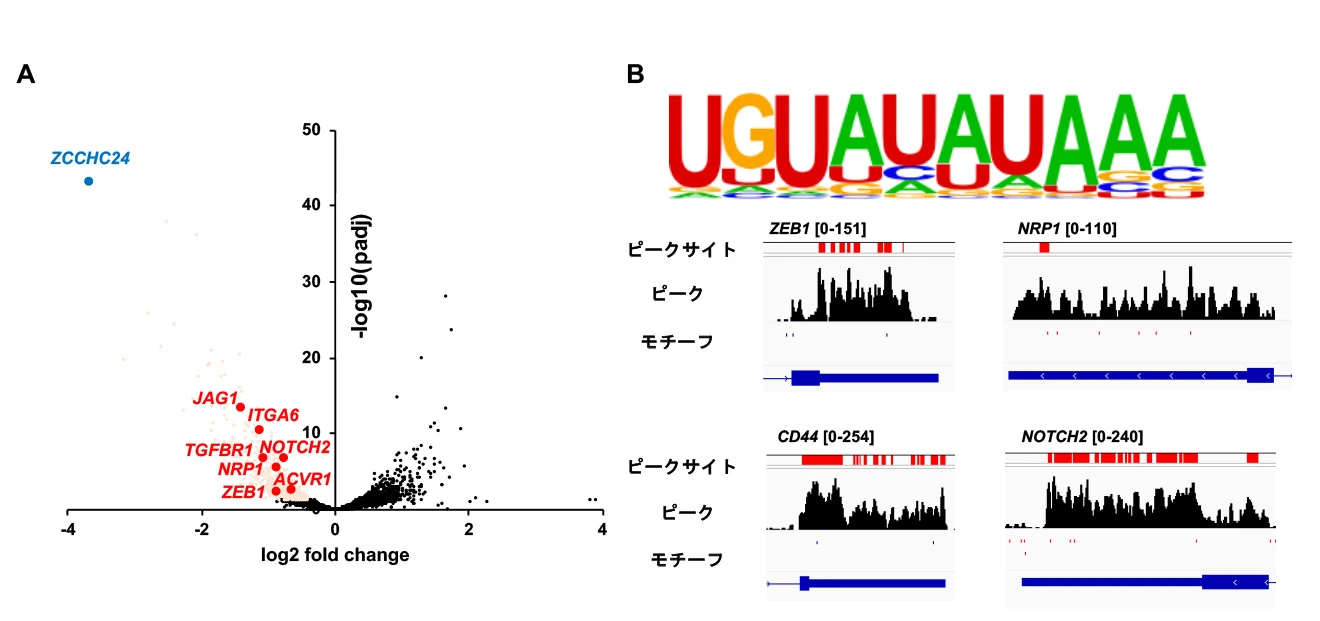
A:ZCCHC24をsiRNAによりノックダウンしたMDAMB231細胞株に対してRNAシークエンスによる網羅的な遺伝子発現解析。B:MDAMB231細胞株内のZCCHC24に対してPAR-CLIPを施行し同定された結合RNAモチーフ配列(上)および標的遺伝子におけるZCCHC24のピーク図(下)。
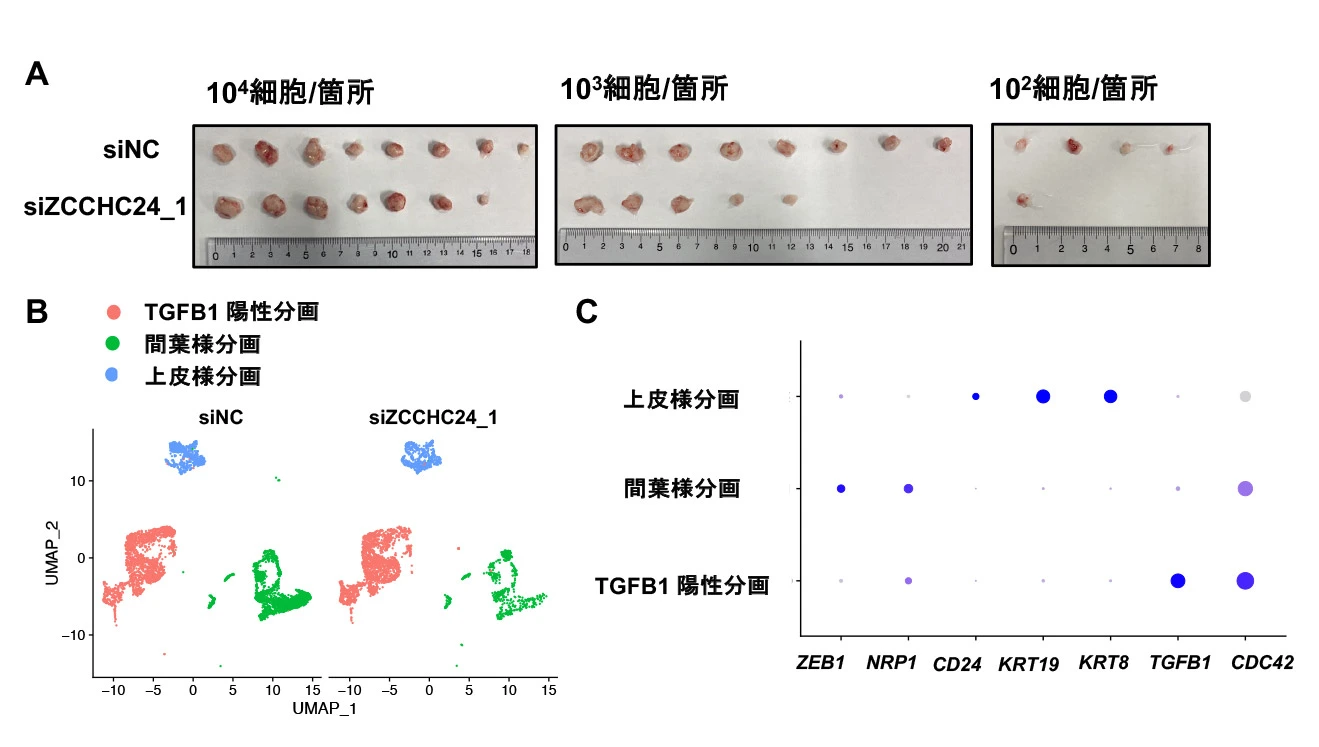
A:ZCCHC24をノックダウンしたTNBC患者由来検体細胞をNOGマウスに皮下移植し、限界希釈法により腫瘍形成能の変化を解析した。B:TNBC患者由来検体細胞に対してZCCHC24およびネガティブコントロール(NC)をノックダウンした状態で1,000個/箇所でNOGマウスに皮下移植し、形成された腫瘍に対してシングルセルRNAシークエンスを行った。図はシングルセルRNAシークエンスに対するDimensional reduction plot (各種類の細胞がどれだけいるのかを簡易的に表す手法)。C:シングルセルRNAシークエンスにおいて各分画にどういった遺伝子が特徴的に発現しているのかを示したDot Plot。
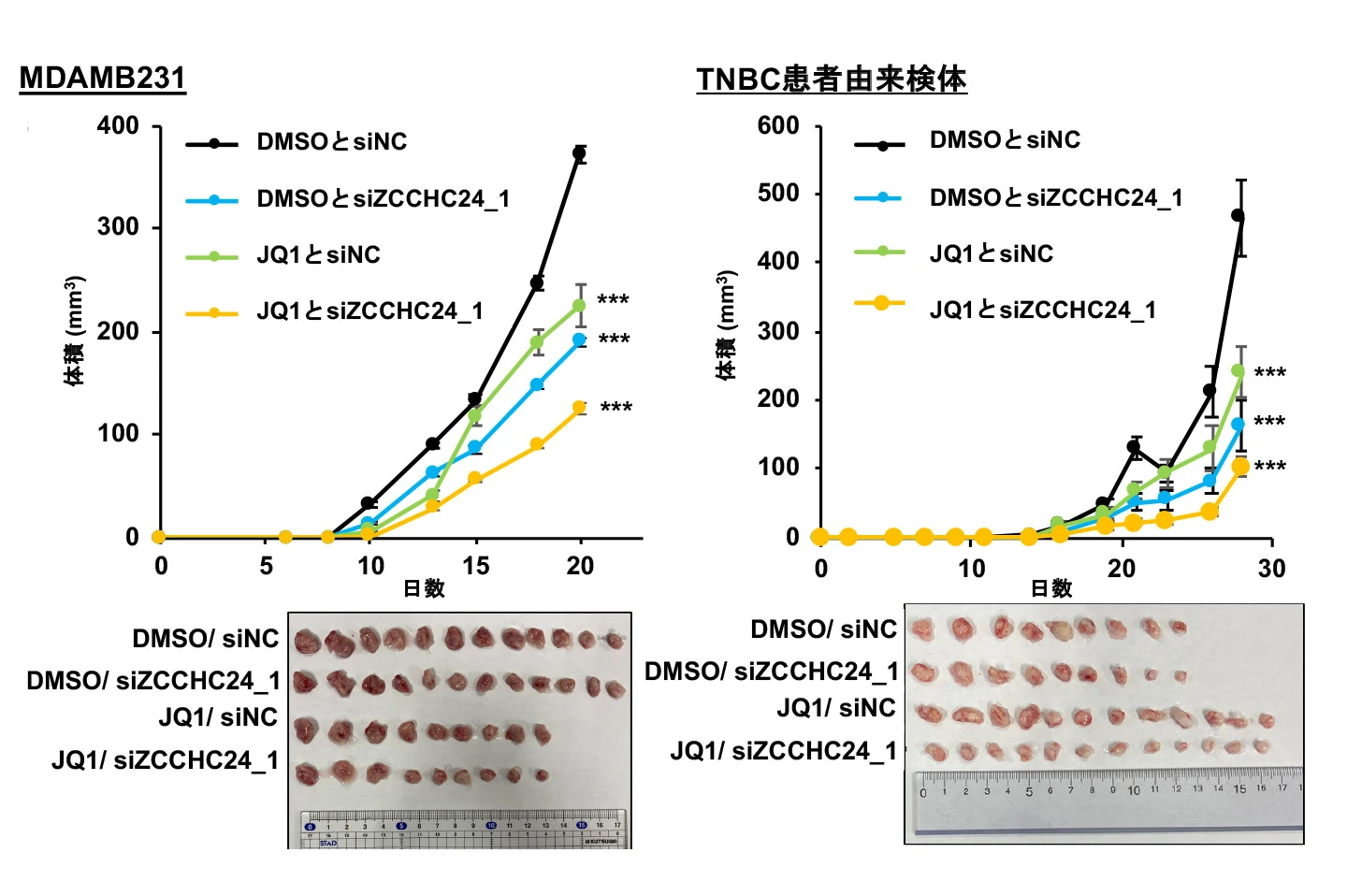
TNBC細胞株MDAMB231およびTNBC患者由来検体に対してZCCHC24およびネガティブコントロール(NC)に対するsiRNAを導入し、免疫不全マウスに皮下移植した。皮下移植されたマウスに対してBET阻害剤およびコントロールとしてDMSOを投与し、腫瘍体積を経時的に計測した。
今後の展開
本研究グループはRNA結合タンパク質ZCCHC24による乳癌幹細胞性に重要な遺伝子の非翻訳領域上の"UGUWHWWA"というcis-elementを介した転写後調節機構と、転写因子ZEB1による転写調節がポジティブフィードバックを形成することでTNBCの腫瘍形成能や化学療法に対する治療抵抗性を獲得するという新規分子メカニズムを明らかにしました(図4)。本研究成果は、乳癌幹細胞制御におけるRNA階層を介したメカニズムの重要性を明らかにするとともに、難治性がんであるTNBCに対する新規治療標的としてZCCHC24が有望な分子であることを示し、今後の乳癌治療への寄与が期待されます。
用語説明
- [用語1]
- トリプルネガティブ乳癌(TNBC):ホルモン療法の標的となるエストロゲン受容体、プロゲステロン受容体、および抗HER2抗体療法の標的となるHER2受容体が陰性の乳癌のこと。治療標的となる受容体がないため、治療抵抗性が高いことで知られる。
- [用語2]
- cis-element:因子が直接結合することで標的遺伝子発現を調節することができる部位の総称。ここではRNA結合タンパク質が直接結合する塩基配列のことで標的mRNAへの機能調節を行う部位のことを指す。
- [用語3]
- シングルセルRNAシークエンス:細胞を性状ごとに分類したり、細胞集団に特異的に発現する遺伝子を同定したりすることを目的として1細胞ごとに網羅的な遺伝子発現解析を行う研究手法。
- [用語4]
- BRICシークエンス:ブロモウリジンを細胞に取り込ませたのちにブロモウリジンに対する抗体により、免疫沈降を行うことによって濃縮されたRNAを計測することにより細胞内におけるmRNAの分解速度を計測する手法。
- [用語5]
- PAR-CLIP法:細胞にチオウリジンを取り込ませた状態で紫外線によりタンパク質とmRNAをクロスリンクさせたのちにRNA結合タンパク質に対して免疫沈降を行うことによって高精度にRNA結合タンパク質の結合標的を網羅的に同定する手法。RNA結合タンパク質の結合モチーフの解析等に適している。
- [用語6]
- Hi-C:DNAが細胞内でどのように三次元的に折り畳まれているのかを網羅的に同定する手法。近接しているDNA上の領域が明らかとなることによって、遺伝子のエンハンサー候補領域の同定などが可能となる。
- [用語7]
- ChIPシークエンス:転写因子とDNAとをホルムアルデヒドによりクロスリンクしたのちに転写因子に対して免疫沈降を行うことによって転写因子が結合しているDNA領域を網羅的に同定する手法。
論文情報
- 掲載誌:
- EMBO Reports
- 論文タイトル:
- RNA binding protein ZCCHC24 promotes tumorigenicity in triple-negative breast cancer
- 著者:
- Yutaro Uchida, Ryota Kurimoto, Tomoki Chiba, Takahide Matsushima, Goshi Oda,Iichiroh Onishi, Yasuto Takeuchi, Noriko Gotoh, Hiroshi Asahara
研究者プロフィール
内田雄太郎 Yutaro UCHIDA
東京科学大学 大学院医歯学総合研究科
システム発生・再生医学分野
博士研究員・臨床研修医(東京科学大学病院)
MDPhDコース卒業生
研究分野:
RNA生物学、核酸医薬学、癌分子生物学

淺原弘嗣 Hiroshi ASAHARA
東京科学大学 大学院医歯学総合研究科
システム発生・再生医学分野 教授
研究分野:
分子生物学(遺伝子発現)、発生・再生医学、整形外科学、リウマチ学

後藤典子 Noriko GOTOH
金沢大学 がん進展制御研究所
分子病態研究分野 教授
研究分野:
分子腫瘍学、幹細胞生物学

関連ページ
お問い合わせ
東京科学大学 大学院医歯学総合研究科 システム発生・再生医学分野
教授 淺原弘嗣
- Tel
- 03-5803-5015
- Fax
- 03-5803-5810
- asahara.syst@tmd.ac.jp
取材申込み
東京科学大学 総務企画部 広報課
- Tel
- 03-5734-2975
- Fax
- 03-5734-3661
- media@ml.tmd.ac.jp
金沢大学薬学・がん研支援課企画総務係
- Tel
- 076-234-6979
- Fax
- 076-234-6844